Un equipo de investigación internacional, liderado por el Instituto de Biología Evolutiva (IBE) en Barcelona, ha construido el catálogo más extenso de diversidad genómica en poblaciones de chimpancés salvajes realizado hasta la fecha.
Como adelantó en su momento Vozpópuli, por primera vez se ha secuenciado la información genómica obtenida de cientos de muestras fecales de chimpancés en toda su área de distribución, aclarando su historia evolutiva y contribuyendo a la conservación de estos grandes simios.
Después de más de tres años de trabajo, el equipo de Tomàs Marquès-Bonet, del Instituto de Biología Evolutiva (IBE) publica los resultados de este nuevo atlas genómico en la revista Cell Genomics, una base de datos que tiene el potencial de crear mapas de rutas y fuentes de tráfico ilegal de chimpancés que podrían usarse para proteger a esta especie en peligro de extinción.
Métodos de ADN antiguo
La información genética se ha recuperado de forma no invasiva utilizando nuevas tecnologías, a partir de cientos de muestras fecales de chimpancés. Por primera vez, se han utilizado los métodos aplicados para analizar el ADN antiguo en poblaciones humanas para recuperar información genética de muestras fecales de grandes simios.
Además, la base de datos genómicos que han desarrollado tiene aplicaciones directas para la conservación de chimpancés, como la identificación de rutas de tráfico ilegal de individuos salvajes y huérfanos.
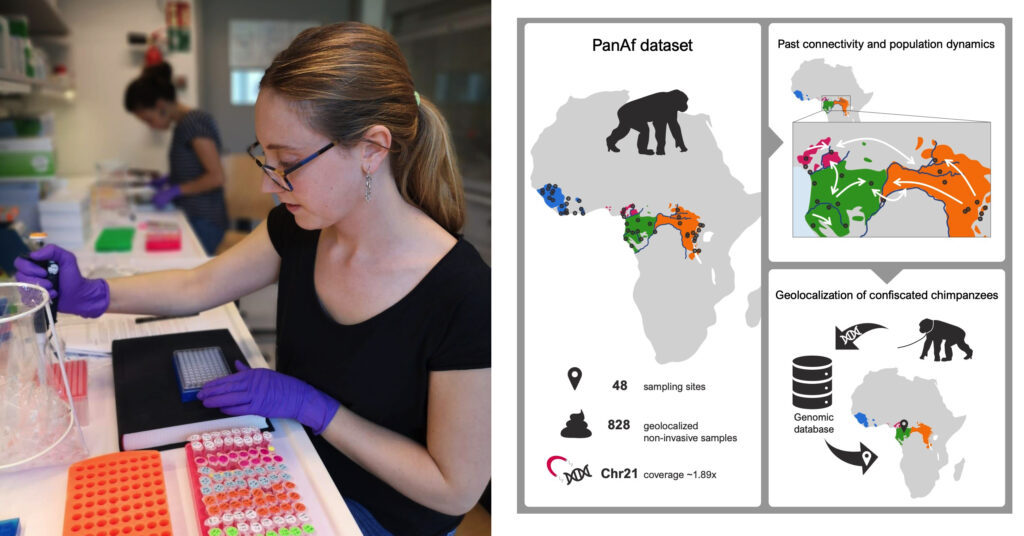
El equipo recuperó información parcial del genoma a partir de más de 800 muestras fecales de chimpancés
El equipo de investigación recuperó información parcial del genoma a partir de más de 800 muestras fecales de chimpancés para crear el atlas de diversidad genómica más grande y detallado de este gran simio africano.
“Usando métodos diseñados para estudiar el ADN antiguo, como en el caso de los neandertales, hemos podido recuperar información genómica de muestras fecales, con las que es muy difícil trabajar. Además, hemos aplicado esa aproximación a un número de muestras de chimpancés sin precedentes”, apunta Marquès-Bonet.
La recolección de muestras no invasivas georreferenciadas de la naturaleza es una forma efectiva de obtener información genómica de chimpancés salvajes cuando muchos factores limitan la recogida de muestras de alta calidad, como sangre o tejidos.
“Hemos visto que las muestras fecales, si bien imponen dificultades técnicas, brindan información genómica muy valiosa para el estudio de las poblaciones de chimpancés salvajes, y también nos permiten la posibilidad de georreferenciarlos y rastrear contactos entre poblaciones sin afectar a su bienestar”, añade Clàudia Fontserè, investigadora del Grupo de Genómica Comparativa del IBE y primera autora del estudio.
Luz sobre su evolución
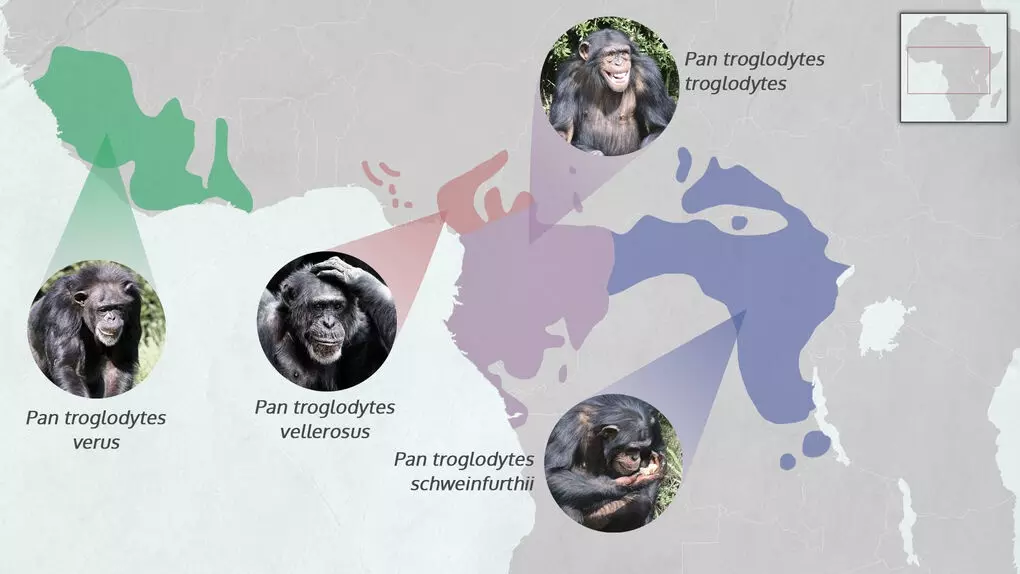
Con este extenso conjunto de datos, los autores arrojan luz sobre el pasado demográfico de los chimpancés y proporcionan nuevas evidencias de la diferenciación genética y los intercambios entre las cuatro subespecies reconocidas.
El equipo de investigación identificó que las características geográficas, como los ríos, constituyen barreras permeables al flujo de genes entre subespecies de chimpancés, pero también entre comunidades. Además, los investigadores propusieron patrones de migración, conectividad y aislamiento entre grupos de chimpancés que han dado forma a la variación del paisaje genómico de estas poblaciones durante los últimos 100.000 años.
“Nuestra aproximación es muy útil para identificar barreras y corredores naturales entre poblaciones"
“Hemos observado que a veces, aunque dos comunidades estén geográficamente muy cerca, puede que vivan a lado y lado de un río y solo hayan tenido contactos muy limitados y esporádicos. Nuestra aproximación es muy útil para identificar barreras y corredores naturales entre poblaciones y puede tener implicaciones para su conservación”, comenta Fontserè.
"Los chimpancés, al igual que los humanos, han tenido una historia evolutiva compleja. Su dinámica y áreas de contacto poblacional pasada y actual deben identificarse claramente para contribuir a la protección de esta especie en peligro de extinción", señala Mimi Arandjelovic, codirectora del estudio e investigadora del Instituto Max Planck de Antropología Evolutiva, el iDiv y la Universidad de Leipzig. Arandjelovic es codirectora del Pan African Programme: The Cultured Chimpanzee (PanAf), un consorcio de investigadores y conservacionistas de África, Europa y América del Norte que pasó ocho años recopilando datos de comportamiento, ecológicos y orgánicos de todo el rango de chimpancés.
Chimpancés incautados
La nueva herramienta genómica ha permitido que el equipo identifique de forma fiable el origen de los individuos, una tarea que hasta ahora no era posible. La capacidad de determinar con precisión el origen de los chimpancés tiene aplicaciones directas para su conservación, como detectar los lugares donde podría concentrarse su caza furtiva e identificar las rutas y orígenes del tráfico ilegal de chimpancés.
“La herramienta desarrollada puede inferir el origen de los chimpancés incautados "
“La herramienta desarrollada puede inferir el origen de los chimpancés incautados, normalmente a unos cientos de kilómetros del origen real, y así proporcionar información fiable sobre las regiones prioritarias a proteger”, añade Marquès-Bonet. La metodología desarrollada ya se está aplicando en proyectos globales de conservación de los otros grandes simios, bonobos, orangutanes y gorilas, como parte de la colaboración con el programa de Illumina iConserve y actualmente ya en proceso de secuenciación en el CNAG-CRG en Barcelona.
Referencia: Population dynamics and genetic connectivity in recent chimpanzee history (Cell Genomics) DOI: https://doi.org/10.1016/j.xgen.2022.100133



Ya no se pueden votar ni publicar comentarios en este artículo.